3D brain tumor segmentation with MONAI
8 minute read
このチュートリアルでは、MONAI を使用してマルチラベル 3D 脳腫瘍セグメンテーションタスクのトレーニング ワークフローを構築し、Weights & Biases の 実験管理 および データ可視化 機能を使用する方法を説明します。このチュートリアルには、次の機能が含まれています。
- Weights & Biases の run を初期化し、再現性のために run に関連付けられたすべての構成を同期します。
- MONAI transform API:
- 辞書形式のデータに対する MONAI Transforms。
- MONAI
transformsAPI に従って新しい transform を定義する方法。 - データ拡張のために強度をランダムに調整する方法。
- データのロードと可視化:
- メタデータ付きの
Nifti画像をロードし、画像のリストをロードしてスタックします。 - トレーニングと検証を高速化するために IO と transform をキャッシュします。
wandb.Tableと Weights & Biases 上のインタラクティブなセグメンテーション オーバーレイを使用してデータを可視化します。
- メタデータ付きの
- 3D
SegResNetモデルのトレーニング- MONAI の
networks、losses、およびmetricsAPI を使用します。 - PyTorch トレーニング ループを使用して 3D
SegResNetモデルをトレーニングします。 - Weights & Biases を使用してトレーニングの experiment を追跡します。
- モデルの チェックポイント を Weights & Biases 上の モデル Artifacts としてログに記録し、バージョン管理します。
- MONAI の
wandb.Tableと Weights & Biases 上のインタラクティブなセグメンテーション オーバーレイを使用して、検証データセットの 予測 を可視化して比較します。
セットアップとインストール
まず、MONAI と Weights & Biases の両方の最新バージョンをインストールします。
!python -c "import monai" || pip install -q -U "monai[nibabel, tqdm]"
!python -c "import wandb" || pip install -q -U wandb
import os
import numpy as np
from tqdm.auto import tqdm
import wandb
from monai.apps import DecathlonDataset
from monai.data import DataLoader, decollate_batch
from monai.losses import DiceLoss
from monai.inferers import sliding_window_inference
from monai.metrics import DiceMetric
from monai.networks.nets import SegResNet
from monai.transforms import (
Activations,
AsDiscrete,
Compose,
LoadImaged,
MapTransform,
NormalizeIntensityd,
Orientationd,
RandFlipd,
RandScaleIntensityd,
RandShiftIntensityd,
RandSpatialCropd,
Spacingd,
EnsureTyped,
EnsureChannelFirstd,
)
from monai.utils import set_determinism
import torch
次に、W&B を使用するために Colab インスタンスを認証します。
wandb.login()
W&B Run の初期化
新しい W&B run を開始して experiment の追跡を開始します。
wandb.init(project="monai-brain-tumor-segmentation")
適切な構成システムの使用は、再現性のある 機械学習 の推奨されるベスト プラクティスです。W&B を使用して、すべての experiment の ハイパーパラメーター を追跡できます。
config = wandb.config
config.seed = 0
config.roi_size = [224, 224, 144]
config.batch_size = 1
config.num_workers = 4
config.max_train_images_visualized = 20
config.max_val_images_visualized = 20
config.dice_loss_smoothen_numerator = 0
config.dice_loss_smoothen_denominator = 1e-5
config.dice_loss_squared_prediction = True
config.dice_loss_target_onehot = False
config.dice_loss_apply_sigmoid = True
config.initial_learning_rate = 1e-4
config.weight_decay = 1e-5
config.max_train_epochs = 50
config.validation_intervals = 1
config.dataset_dir = "./dataset/"
config.checkpoint_dir = "./checkpoints"
config.inference_roi_size = (128, 128, 64)
config.max_prediction_images_visualized = 20
また、モジュールのランダム シードを設定して、確定的トレーニングを有効または無効にする必要もあります。
set_determinism(seed=config.seed)
# ディレクトリを作成する
os.makedirs(config.dataset_dir, exist_ok=True)
os.makedirs(config.checkpoint_dir, exist_ok=True)
データのロードと変換
ここでは、monai.transforms API を使用して、マルチクラス ラベルを one-hot 形式のマルチラベル セグメンテーション タスクに変換するカスタム transform を作成します。
class ConvertToMultiChannelBasedOnBratsClassesd(MapTransform):
"""
brats クラスに基づいてラベルをマルチ チャンネルに変換します:
label 1 は周囲浮腫です
label 2 は GD 増強腫瘍です
label 3 は壊死性および非増強性腫瘍コアです
可能なクラスは TC (腫瘍コア)、WT (全腫瘍) および ET (増強腫瘍) です。
参考: https://github.com/Project-MONAI/tutorials/blob/main/3d_segmentation/brats_segmentation_3d.ipynb
"""
def __call__(self, data):
d = dict(data)
for key in self.keys:
result = []
# label 2 と label 3 をマージして TC を構築する
result.append(torch.logical_or(d[key] == 2, d[key] == 3))
# labels 1、2、3 をマージして WT を構築する
result.append(
torch.logical_or(
torch.logical_or(d[key] == 2, d[key] == 3), d[key] == 1
)
)
# label 2 は ET です
result.append(d[key] == 2)
d[key] = torch.stack(result, axis=0).float()
return d
次に、トレーニング データセットと検証データセットの transform をそれぞれ設定します。
train_transform = Compose(
[
# 4 つの Nifti 画像をロードして一緒にスタックする
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys="image"),
EnsureTyped(keys=["image", "label"]),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(
keys=["image", "label"],
pixdim=(1.0, 1.0, 1.0),
mode=("bilinear", "nearest"),
),
RandSpatialCropd(
keys=["image", "label"], roi_size=config.roi_size, random_size=False
),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=0),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=1),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=2),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
RandScaleIntensityd(keys="image", factors=0.1, prob=1.0),
RandShiftIntensityd(keys="image", offsets=0.1, prob=1.0),
]
)
val_transform = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys="image"),
EnsureTyped(keys=["image", "label"]),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(
keys=["image", "label"],
pixdim=(1.0, 1.0, 1.0),
mode=("bilinear", "nearest"),
),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
]
)
データセット
この experiment に使用される データセット は、http://medicaldecathlon.com/ から提供されています。マルチモーダル マルチサイト MRI データ (FLAIR、T1w、T1gd、T2w) を使用して、グリオーマ、壊死/活性腫瘍、および浮腫をセグメント化します。データセット は、750 個の 4D ボリューム (484 トレーニング + 266 テスト) で構成されています。
DecathlonDataset を使用して、 データセット を自動的にダウンロードして抽出します。これは、MONAI CacheDataset を継承しており、cache_num=N を設定してトレーニング用に N 個のアイテムをキャッシュし、メモリ サイズに応じて、デフォルトの 引数 を使用して検証用にすべてのアイテムをキャッシュできます。
train_dataset = DecathlonDataset(
root_dir=config.dataset_dir,
task="Task01_BrainTumour",
transform=val_transform,
section="training",
download=True,
cache_rate=0.0,
num_workers=4,
)
val_dataset = DecathlonDataset(
root_dir=config.dataset_dir,
task="Task01_BrainTumour",
transform=val_transform,
section="validation",
download=False,
cache_rate=0.0,
num_workers=4,
)
train_transform を train_dataset に適用する代わりに、val_transform をトレーニング データセット と 検証 データセット の両方に適用します。これは、トレーニングの前に、 データセット の両方の分割から サンプル を 可視化 するためです。データセット の可視化
Weights & Biases は、画像、ビデオ、オーディオなどをサポートしています。リッチ メディアをログに記録して 結果 を調査し、run、モデル、および データセット を視覚的に比較できます。セグメンテーション マスク オーバーレイ システム を使用して、データ ボリュームを可視化します。Tables でセグメンテーション マスクをログに記録するには、テーブル の各行に wandb.Image オブジェクトを指定する必要があります。
次に、疑似コードの例を示します。
table = wandb.Table(columns=["ID", "Image"])
for id, img, label in zip(ids, images, labels):
mask_img = wandb.Image(
img,
masks={
"prediction": {"mask_data": label, "class_labels": class_labels}
# ...
},
)
table.add_data(id, img)
wandb.log({"Table": table})
次に、サンプル画像、ラベル、wandb.Table オブジェクト、および関連するメタデータを受け取り、Weights & Biases ダッシュボードにログに記録されるテーブル の行に入力する簡単なユーティリティ関数を作成します。
def log_data_samples_into_tables(
sample_image: np.array,
sample_label: np.array,
split: str = None,
data_idx: int = None,
table: wandb.Table = None,
):
num_channels, _, _, num_slices = sample_image.shape
with tqdm(total=num_slices, leave=False) as progress_bar:
for slice_idx in range(num_slices):
ground_truth_wandb_images = []
for channel_idx in range(num_channels):
ground_truth_wandb_images.append(
masks = {
"ground-truth/Tumor-Core": {
"mask_data": sample_label[0, :, :, slice_idx],
"class_labels": {0: "background", 1: "Tumor Core"},
},
"ground-truth/Whole-Tumor": {
"mask_data": sample_label[1, :, :, slice_idx] * 2,
"class_labels": {0: "background", 2: "Whole Tumor"},
},
"ground-truth/Enhancing-Tumor": {
"mask_data": sample_label[2, :, :, slice_idx] * 3,
"class_labels": {0: "background", 3: "Enhancing Tumor"},
},
}
wandb.Image(
sample_image[channel_idx, :, :, slice_idx],
masks=masks,
)
)
table.add_data(split, data_idx, slice_idx, *ground_truth_wandb_images)
progress_bar.update(1)
return table
次に、wandb.Table オブジェクトと、データ可視化で入力できるようにするために構成される 列 を定義します。
table = wandb.Table(
columns=[
"Split",
"Data Index",
"Slice Index",
"Image-Channel-0",
"Image-Channel-1",
"Image-Channel-2",
"Image-Channel-3",
]
)
次に、train_dataset と val_dataset をそれぞれループして、データ サンプル の 可視化 を生成し、ダッシュボード にログに記録するテーブル の行に入力します。
# train_dataset の 可視化 を生成する
max_samples = (
min(config.max_train_images_visualized, len(train_dataset))
if config.max_train_images_visualized > 0
else len(train_dataset)
)
progress_bar = tqdm(
enumerate(train_dataset[:max_samples]),
total=max_samples,
desc="トレーニング データセット の 可視化 を生成しています:",
)
for data_idx, sample in progress_bar:
sample_image = sample["image"].detach().cpu().numpy()
sample_label = sample["label"].detach().cpu().numpy()
table = log_data_samples_into_tables(
sample_image,
sample_label,
split="train",
data_idx=data_idx,
table=table,
)
# val_dataset の 可視化 を生成する
max_samples = (
min(config.max_val_images_visualized, len(val_dataset))
if config.max_val_images_visualized > 0
else len(val_dataset)
)
progress_bar = tqdm(
enumerate(val_dataset[:max_samples]),
total=max_samples,
desc="検証 データセット の 可視化 を生成しています:",
)
for data_idx, sample in progress_bar:
sample_image = sample["image"].detach().cpu().numpy()
sample_label = sample["label"].detach().cpu().numpy()
table = log_data_samples_into_tables(
sample_image,
sample_label,
split="val",
data_idx=data_idx,
table=table,
)
# テーブル をダッシュボードにログに記録する
wandb.log({"Tumor-Segmentation-Data": table})
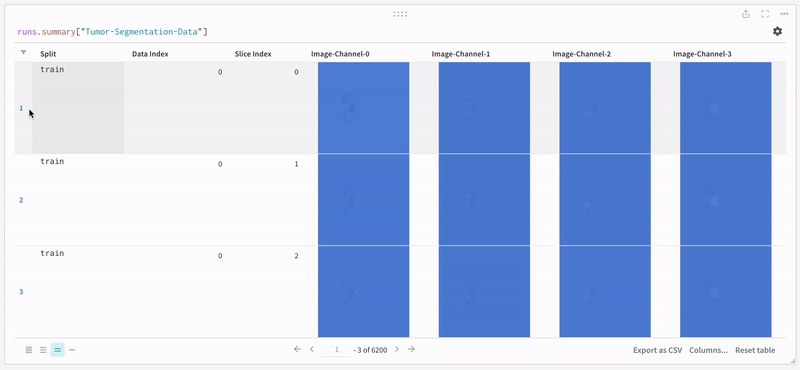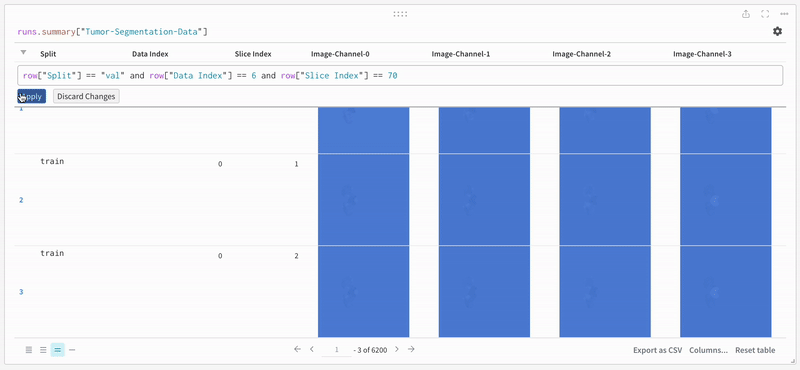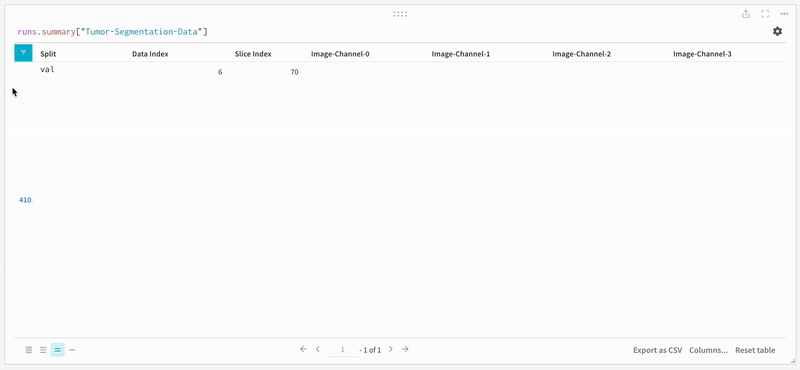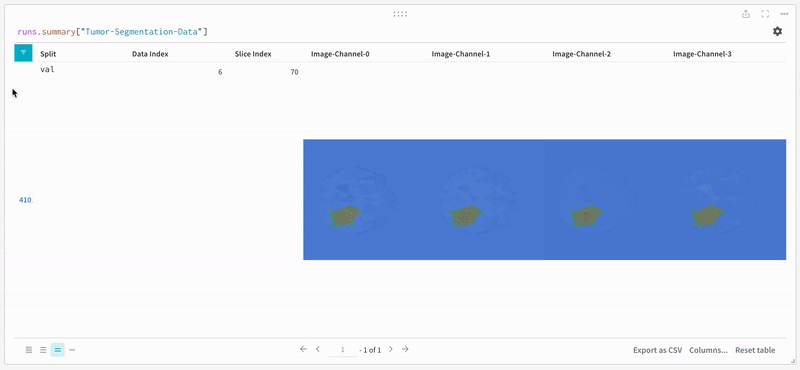
データ は、インタラクティブなテーブル形式で W&B ダッシュボード に表示されます。各行にそれぞれのセグメンテーション マスク がオーバーレイされたデータ ボリュームの特定の スライス の各 チャンネル を確認できます。Weave クエリ を記述して、テーブル のデータをフィルタリングし、特定の 1 つの行に焦点を当てることができます。
 |
|---|
| ログに記録されたテーブル データの例。 |
画像を開き、インタラクティブなオーバーレイを使用して各セグメンテーション マスク を操作する方法を確認します。
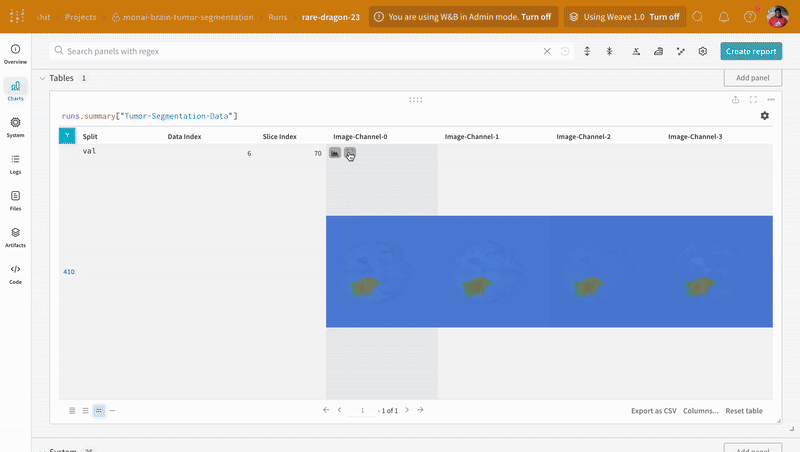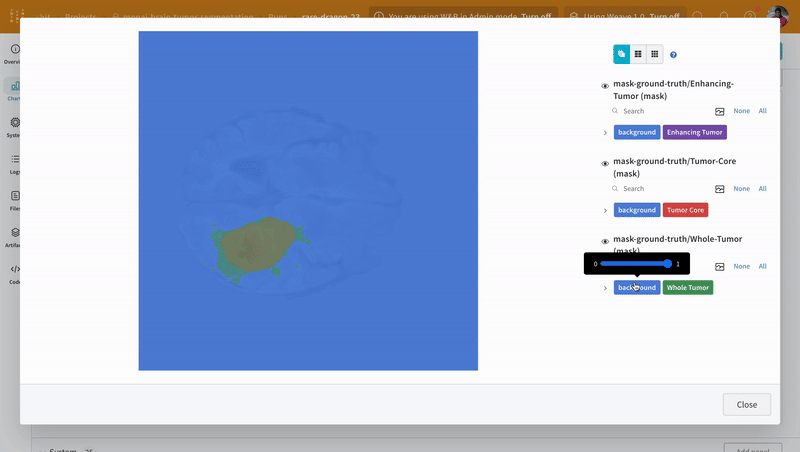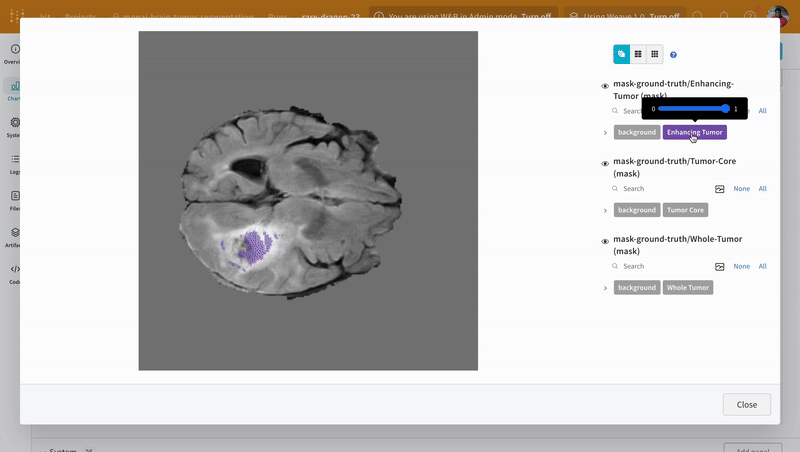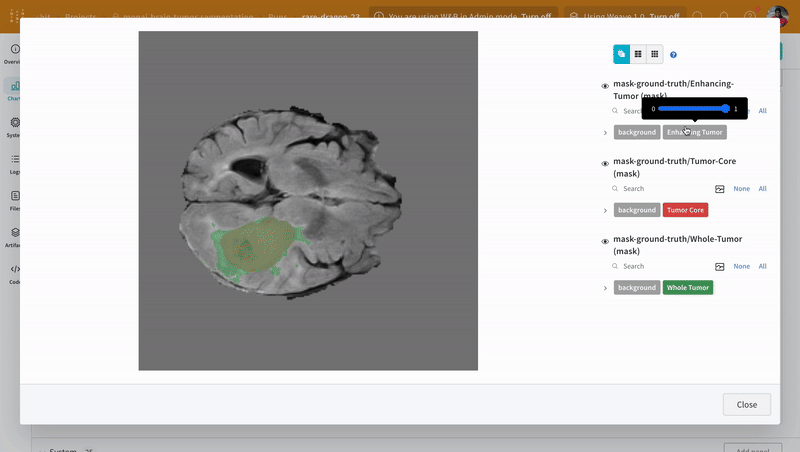 |
|---|
| 可視化されたセグメンテーション マップ の例。 |
データのロード
データセット からデータをロードするために、PyTorch DataLoaders を作成します。DataLoaders を作成する前に、トレーニング用にデータを事前処理および変換するために、train_dataset の transform を train_transform に設定します。
# train_transforms をトレーニング データセット に適用する
train_dataset.transform = train_transform
# train_loader を作成する
train_loader = DataLoader(
train_dataset,
batch_size=config.batch_size,
shuffle=True,
num_workers=config.num_workers,
)
# val_loader を作成する
val_loader = DataLoader(
val_dataset,
batch_size=config.batch_size,
shuffle=False,
num_workers=config.num_workers,
)
モデル、損失、および オプティマイザー の作成
このチュートリアルでは、論文 自動エンコーダー正則化を使用した 3D MRI 脳腫瘍セグメンテーション に基づいて SegResNet モデルを作成します。SegResNet モデルは、monai.networks API の一部として PyTorch モジュールとして実装され、 オプティマイザー と 学習率 スケジューラ も実装されています。
device = torch.device("cuda:0")
# モデルを作成する
model = SegResNet(
blocks_down=[1, 2, 2, 4],
blocks_up=[1, 1, 1],
init_filters=16,
in_channels=4,
out_channels=3,
dropout_prob=0.2,
).to(device)
# オプティマイザー を作成する
optimizer = torch.optim.Adam(
model.parameters(),
config.initial_learning_rate,
weight_decay=config.weight_decay,
)
# 学習率 スケジューラ を作成する
lr_scheduler = torch.optim.lr_scheduler.CosineAnnealingLR(
optimizer, T_max=config.max_train_epochs
)
monai.losses API を使用して損失をマルチラベル DiceLoss として定義し、monai.metrics API を使用して対応する dice メトリクスを定義します。
loss_function = DiceLoss(
smooth_nr=config.dice_loss_smoothen_numerator,
smooth_dr=config.dice_loss_smoothen_denominator,
squared_pred=config.dice_loss_squared_prediction,
to_onehot_y=config.dice_loss_target_onehot,
sigmoid=config.dice_loss_apply_sigmoid,
)
dice_metric = DiceMetric(include_background=True, reduction="mean")
dice_metric_batch = DiceMetric(include_background=True, reduction="mean_batch")
post_trans = Compose([Activations(sigmoid=True), AsDiscrete(threshold=0.5)])
# 自動混合精度を使用してトレーニングを高速化する
scaler = torch.cuda.amp.GradScaler()
torch.backends.cudnn.benchmark = True
混合精度推論用の小さなユーティリティを定義します。これは、トレーニング プロセスの検証ステップ中、およびトレーニング後にモデルを実行する場合に役立ちます。
def inference(model, input):
def _compute(input):
return sliding_window_inference(
inputs=input,
roi_size=(240, 240, 160),
sw_batch_size=1,
predictor=model,
overlap=0.5,
)
with torch.cuda.amp.autocast():
return _compute(input)
トレーニングと検証
トレーニングの前に、トレーニング および 検証 experiment を追跡するために、後で wandb.log() でログに記録される メトリクス プロパティを定義します。
wandb.define_metric("epoch/epoch_step")
wandb.define_metric("epoch/*", step_metric="epoch/epoch_step")
wandb.define_metric("batch/batch_step")
wandb.define_metric("batch/*", step_metric="batch/batch_step")
wandb.define_metric("validation/validation_step")
wandb.define_metric("validation/*", step_metric="validation/validation_step")
batch_step = 0
validation_step = 0
metric_values = []
metric_values_tumor_core = []
metric_values_whole_tumor = []
metric_values_enhanced_tumor = []
標準的な PyTorch トレーニング ループを実行する
# W&B Artifact オブジェクトを定義する
artifact = wandb.Artifact(
name=f"{wandb.run.id}-checkpoint", type="model"
)
epoch_progress_bar = tqdm(range(config.max_train_epochs), desc="トレーニング:")
for epoch in epoch_progress_bar:
model.train()
epoch_loss = 0
total_batch_steps = len(train_dataset) // train_loader.batch_size
batch_progress_bar = tqdm(train_loader, total=total_batch_steps, leave=False)
# トレーニング ステップ
for batch_data in batch_progress_bar:
inputs, labels = (
batch_data["image"].to(device),
batch_data["label"].to(device),
)
optimizer.zero_grad()
with torch.cuda.amp.autocast():
outputs = model(inputs)
loss = loss_function(outputs, labels)
scaler.scale(loss).backward()
scaler.step(optimizer)
scaler.update()
epoch_loss += loss.item()
batch_progress_bar.set_description(f"train_loss: {loss.item():.4f}:")
## バッチごとのトレーニング損失を W&B にログに記録する
wandb.log({"batch/batch_step": batch_step, "batch/train_loss": loss.item()})
batch_step += 1
lr_scheduler.step()
epoch_loss /= total_batch_steps
## バッチごとのトレーニング損失と 学習率 を W&B にログに記録する
wandb.log(
{
"epoch/epoch_step": epoch,
"epoch/mean_train_loss": epoch_loss,
"epoch/learning_rate": lr_scheduler.get_last_lr()[0],
}
)
epoch_progress_bar.set_description(f"トレーニング: train_loss: {epoch_loss:.4f}:")
# 検証と モデル チェックポイント ステップ
if (epoch + 1) % config.validation_intervals == 0:
model.eval()
with torch.no_grad():
for val_data in val_loader:
val_inputs, val_labels = (
val_data["image"].to(device),
val_data["label"].to(device),
)
val_outputs = inference(model, val_inputs)
val_outputs = [post_trans(i) for i in decollate_batch(val_outputs)]
dice_metric(y_pred=val_outputs, y=val_labels)
dice_metric_batch(y_pred=val_outputs, y=val_labels)
metric_values.append(dice_metric.aggregate().item())
metric_batch = dice_metric_batch.aggregate()
metric_values_tumor_core.append(metric_batch[0].item())
metric_values_whole_tumor.append(metric_batch[1].item())
metric_values_enhanced_tumor.append(metric_batch[2].item())
dice_metric.reset()
dice_metric_batch.reset()
checkpoint_path = os.path.join(config.checkpoint_dir, "model.pth")
torch.save(model.state_dict(), checkpoint_path)
# W&B Artifacts を使用して モデル チェックポイント をログに記録し、バージョン管理する。
artifact.add_file(local_path=checkpoint_path)
wandb.log_artifact(artifact, aliases=[f"epoch_{epoch}"])
# 検証 メトリクス を W&B ダッシュボード にログに記録する。
wandb.log(
{
"validation/validation_step": validation_step,
"validation/mean_dice": metric_values[-1],
"validation/mean_dice_tumor_core": metric_values_tumor_core[-1],
"validation/mean_dice_whole_tumor": metric_values_whole_tumor[-1],
"validation/mean_dice_enhanced_tumor": metric_values_enhanced_tumor[-1],
}
)
validation_step += 1
# この Artifact がログ記録を完了するまで待機する
artifact.wait()
wandb.log でコードをインストルメント化すると、トレーニング および 検証 プロセスに関連付けられたすべての メトリクス だけでなく、W&B ダッシュボード 上のすべてのシステム メトリクス (この場合は CPU と GPU) も追跡できます。
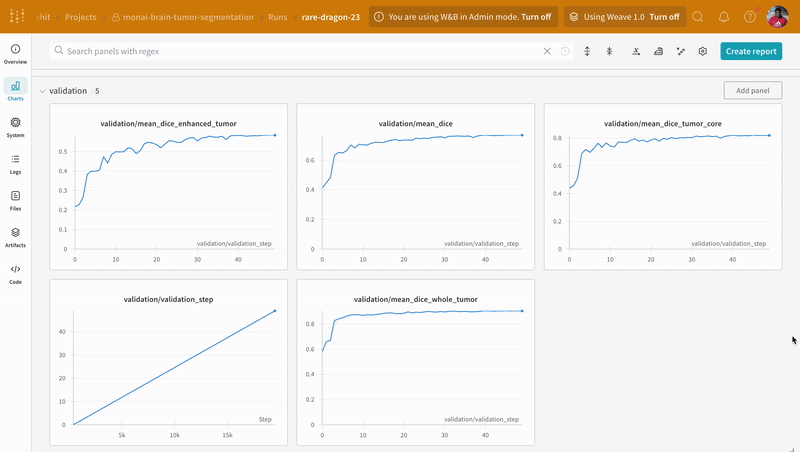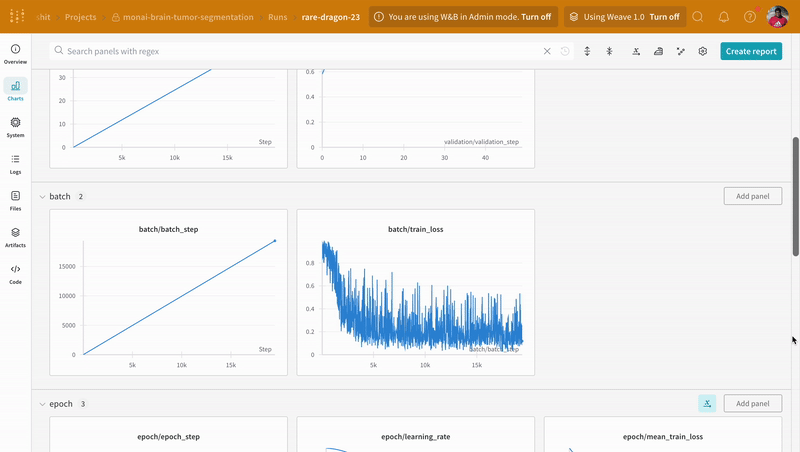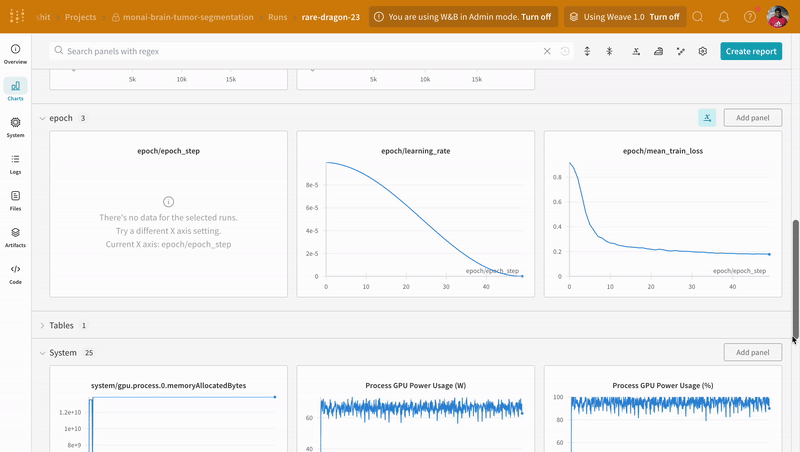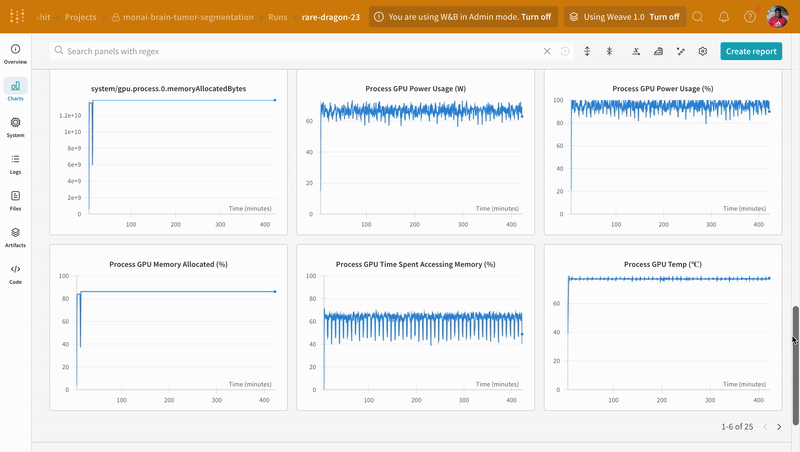 |
|---|
| W&B でのトレーニング および 検証 プロセスの追跡の例。 |
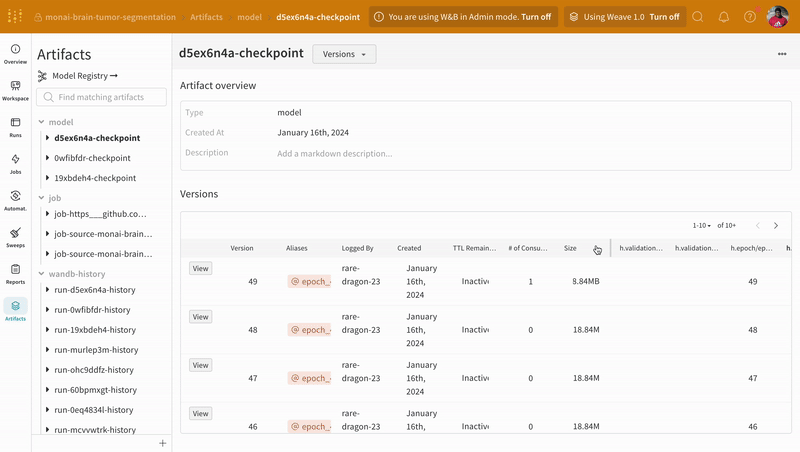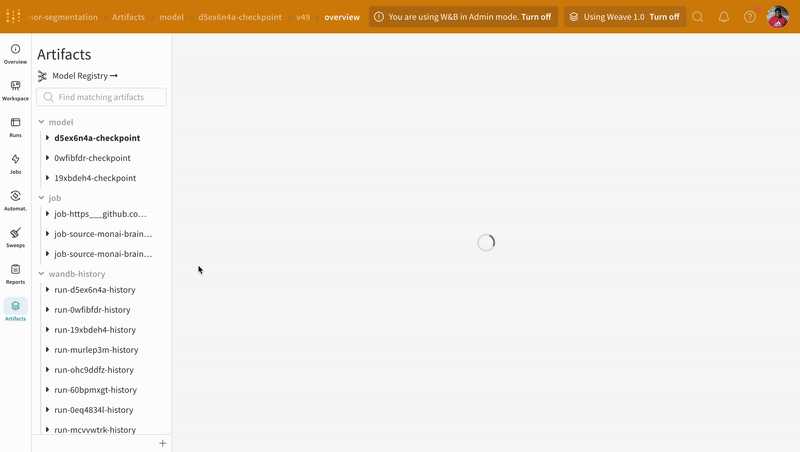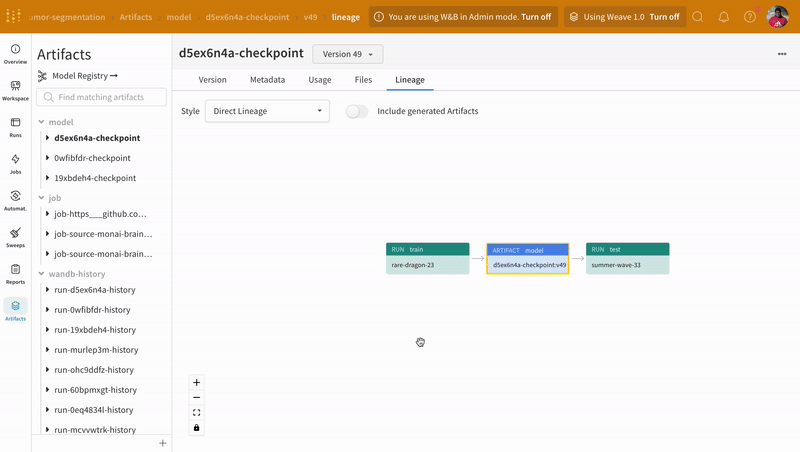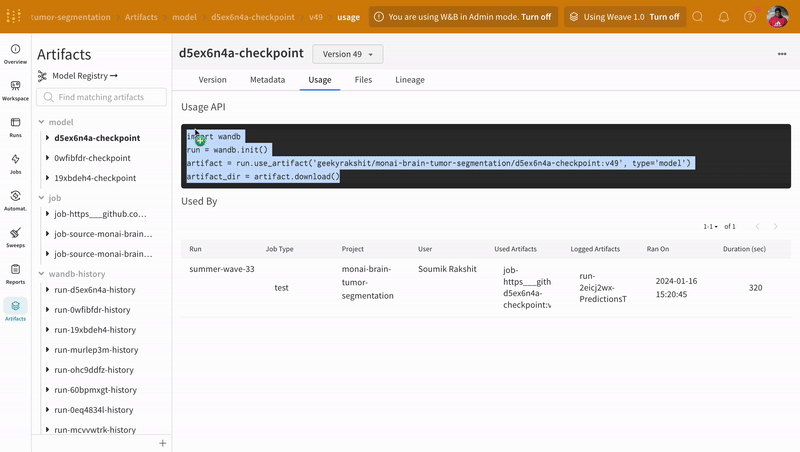
W&B run ダッシュボード の Artifacts タブに移動して、トレーニング 中にログに記録された モデル チェックポイント Artifacts のさまざまな バージョン にアクセスします。
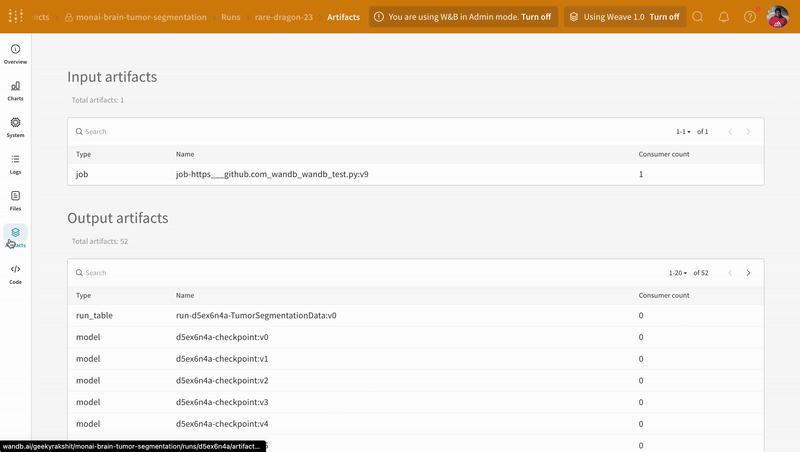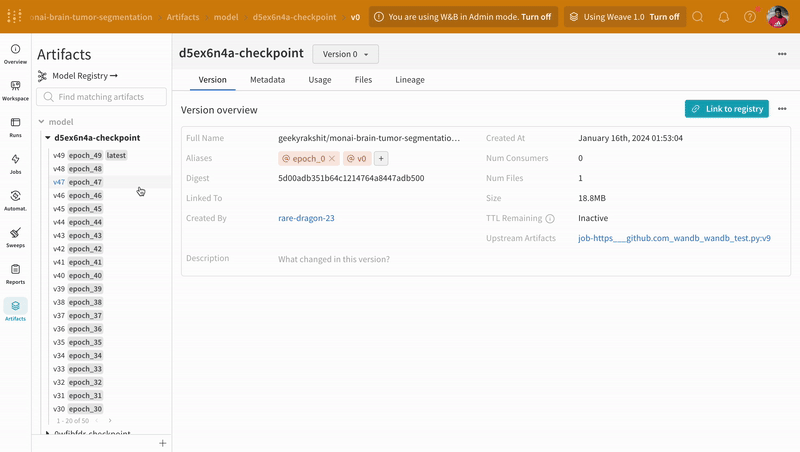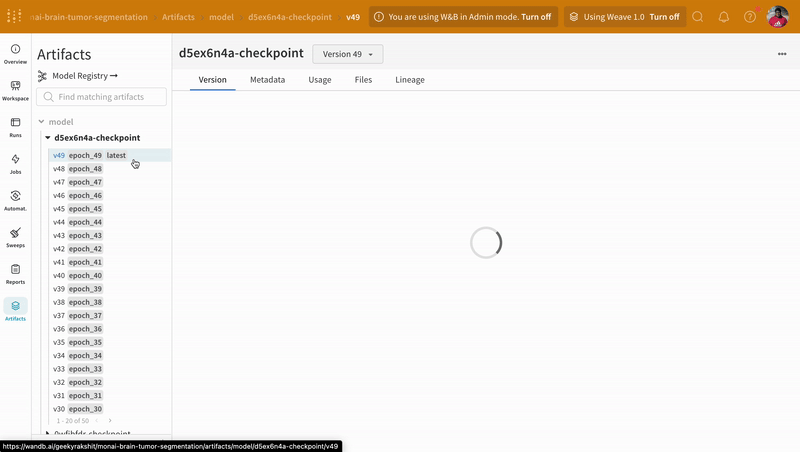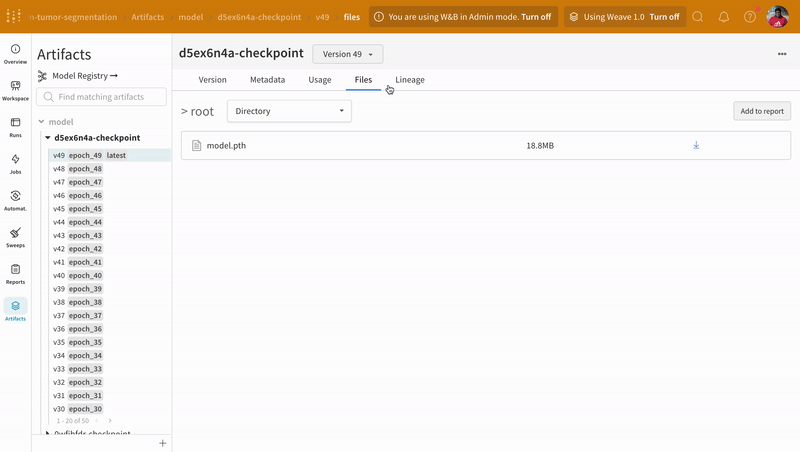 |
|---|
| W&B での モデル チェックポイント のログ記録と バージョン管理 の例。 |
推論
Artifacts インターフェイスを使用すると、最適な モデル チェックポイント である Artifact の バージョン を選択できます。この場合、エポックごとの平均トレーニング損失です。Artifact のリネージ全体を調べて、必要な バージョン を使用することもできます。
 |
|---|
| W&B での モデル Artifact の追跡の例。 |
エポックごとの平均トレーニング損失が最適なモデル Artifact の バージョン をフェッチし、チェックポイント 状態 辞書 をモデルにロードします。
model_artifact = wandb.use_artifact(
"geekyrakshit/monai-brain-tumor-segmentation/d5ex6n4a-checkpoint:v49",
type="model",
)
model_artifact_dir = model_artifact.download()
model.load_state_dict(torch.load(os.path.join(model_artifact_dir, "model.pth")))
model.eval()
予測 の可視化と 正解 ラベルとの比較
インタラクティブなセグメンテーション マスク オーバーレイを使用して、学習済み モデル の 予測 を 可視化 し、対応する 正解 セグメンテーション マスク と比較するための別のユーティリティ関数を作成します。
def log_predictions_into_tables(
sample_image: np.array,
sample_label: np.array,
predicted_label: np.array,
split: str = None,
data_idx: int = None,
table: wandb.Table = None,
):
num_channels, _, _, num_slices = sample_image.shape
with tqdm(total=num_slices, leave=False) as progress_bar:
for slice_idx in range(num_slices):
wandb_images = []
for channel_idx in range(num_channels):
wandb_images += [
wandb.Image(
sample_image[channel_idx, :, :, slice_idx],
masks={
"ground-truth/Tumor-Core": {
"mask_data": sample_label[0, :, :, slice_idx],
"class_labels": {0: "background", 1: "Tumor Core"},
},
"prediction/Tumor-Core": {
"mask_data": predicted_label[0, :, :, slice_idx] * 2,
"class_labels": {0: "background", 2: "Tumor Core"},
},
},
),
wandb.Image(
sample_image[channel_idx, :, :, slice_idx],
masks={
"ground-truth/Whole-Tumor": {
"mask_data": sample_label[1, :, :, slice_idx],
"class_labels": {0: "background", 1: "Whole Tumor"},
},
"prediction/Whole-Tumor": {
"mask_data": predicted_label[1, :, :, slice_idx] * 2,
"class_labels": {0: "background", 2: "Whole Tumor"},
},
},
),
wandb.Image(
sample_image[channel_idx, :, :, slice_idx],
masks={
"ground-truth/Enhancing-Tumor": {
"mask_data": sample_label[2, :, :, slice_idx],
"class_labels": {0: "background", 1: "Enhancing Tumor"},
},
"prediction/Enhancing-Tumor": {
"mask_data": predicted_label[2, :, :, slice_idx] * 2,
"class_labels": {0: "background", 2: "Enhancing Tumor"},
},
},
),
]
table.add_data(split, data_idx, slice_idx, *wandb_images)
progress_bar.update(1)
return table
予測 結果を 予測 テーブル にログに記録します。
# 予測 テーブル を作成する
prediction_table = wandb.Table(
columns=[
"Split",
"Data Index",
"Slice Index",
"Image-Channel-0/Tumor-Core",
"Image-Channel-1/Tumor-Core",
"Image-Channel-2/Tumor-Core",
"Image-Channel-3/Tumor-Core",
"Image-Channel-0/Whole-Tumor",
"Image-Channel-1/Whole-Tumor",
"Image-Channel-2/Whole-Tumor",
"Image-Channel-3/Whole-Tumor",
"Image-Channel-0/Enhancing-Tumor",
"Image-Channel-1/Enhancing-Tumor",
"Image-Channel-2/Enhancing-Tumor",
"Image-Channel-3/Enhancing-Tumor",
]
)
# 推論 と 可視化 を実行する
with torch.no_grad():
config.max_prediction_images_visualized
max_samples = (
min(config.max_prediction_images_visualized, len(val_dataset))
if config.max_prediction_images_visualized > 0
else len(val_dataset)
)
progress_bar = tqdm(
enumerate(val_dataset[:max_samples]),
total=max_samples,
desc="予測 を生成しています:",
)
for data_idx, sample in progress_bar:
val_input = sample["image"].unsqueeze(0).to(device)
val_output = inference(model, val_input)
val_output = post_trans(val_output[0])
prediction_table = log_predictions_into_tables(
sample_image=sample["image"].cpu().numpy(),
sample_label=sample["label"].cpu().numpy(),
predicted_label=val_output.cpu().numpy(),
data_idx=data_idx,
split="validation",
table=prediction_table,
)
wandb.log({"Predictions/Tumor-Segmentation-Data": prediction_table})
# experiment を終了する
wandb.finish()
インタラクティブなセグメンテーション マスク オーバーレイを使用して、各クラスの 予測 されたセグメンテーション マスク と 正解 ラベルを分析および比較します。
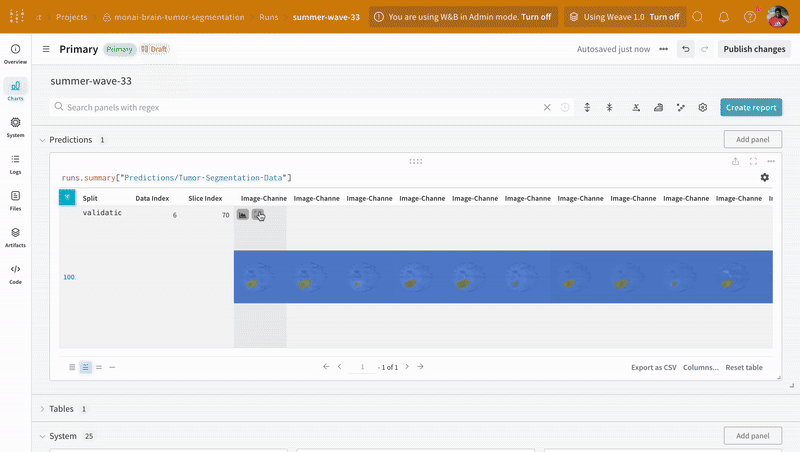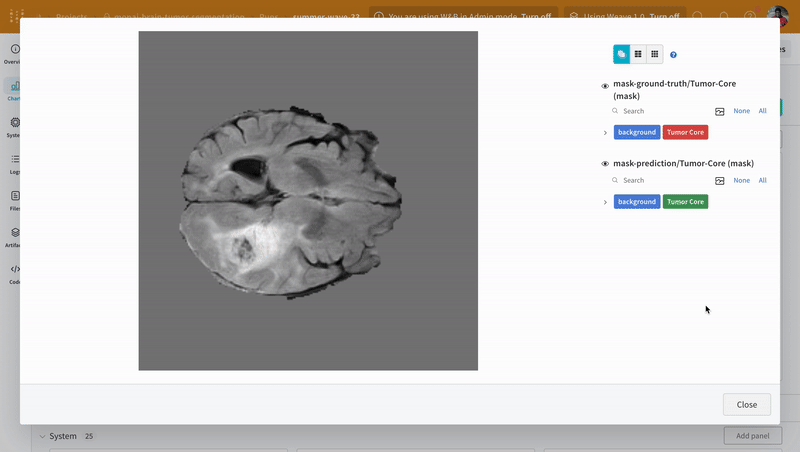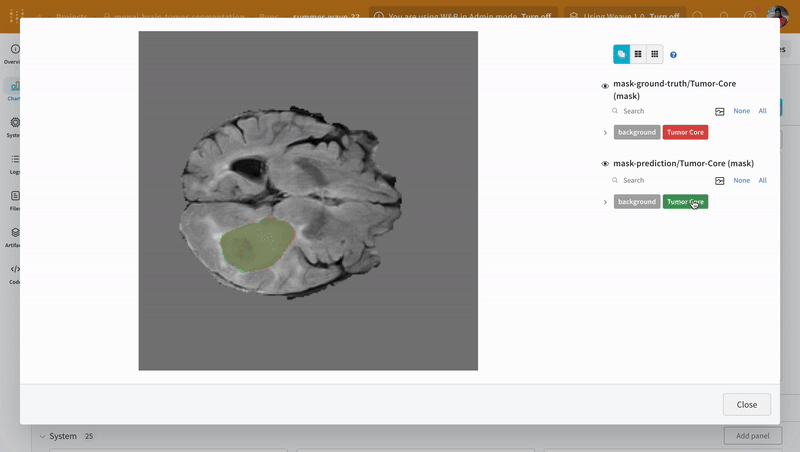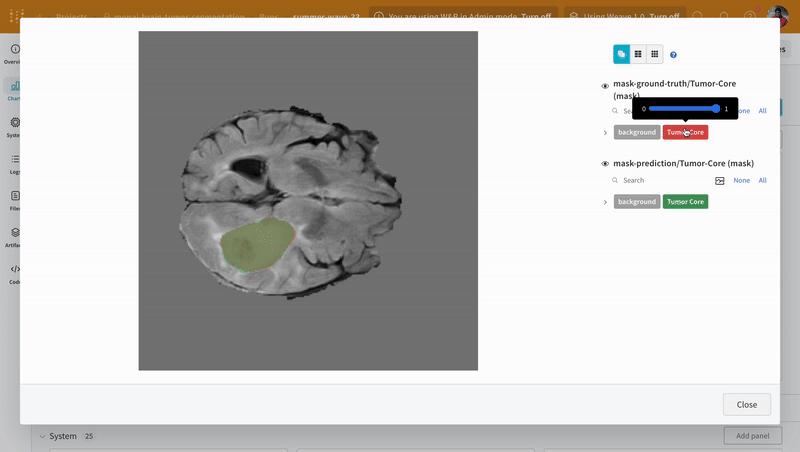 |
|---|
| W&B での 予測 と 正解 の 可視化 の例。 |
謝辞とその他のリソース
[i18n] feedback_title
[i18n] feedback_question
Glad to hear it! Please tell us how we can improve.
Sorry to hear that. Please tell us how we can improve.