3D brain tumor segmentation with MONAI
10 minute read
이 튜토리얼에서는 MONAI를 사용하여 다중 레이블 3D 뇌종양 분할 작업의 트레이닝 워크플로우를 구성하고 Weights & Biases의 실험 추적 및 데이터 시각화 기능을 사용하는 방법을 보여줍니다. 이 튜토리얼에는 다음과 같은 기능이 포함되어 있습니다.
- Weights & Biases run을 초기화하고 재현성을 위해 run과 관련된 모든 구성을 동기화합니다.
- MONAI transform API:
- 사전 형식 데이터에 대한 MONAI Transforms.
- MONAI
transformsAPI에 따라 새로운 transform을 정의하는 방법. - 데이터 증강을 위해 강도를 임의로 조정하는 방법.
- 데이터 로딩 및 시각화:
- 메타데이터와 함께
Nifti이미지를 로드하고, 이미지 목록을 로드하고 스택합니다. - 트레이닝 및 유효성 검사를 가속화하기 위해 IO 및 transforms를 캐시합니다.
wandb.Table및 Weights & Biases의 대화형 분할 오버레이를 사용하여 데이터를 시각화합니다.
- 메타데이터와 함께
- 3D
SegResNet모델 트레이닝- MONAI의
networks,losses및metricsAPI를 사용합니다. - PyTorch 트레이닝 루프를 사용하여 3D
SegResNet모델을 트레이닝합니다. - Weights & Biases를 사용하여 트레이닝 실험을 추적합니다.
- Weights & Biases에서 모델 체크포인트를 모델 Artifacts로 로그하고 버전을 관리합니다.
- MONAI의
wandb.Table및 Weights & Biases의 대화형 분할 오버레이를 사용하여 유효성 검사 데이터셋에서 예측을 시각화하고 비교합니다.
설정 및 설치
먼저 MONAI와 Weights & Biases의 최신 버전을 설치합니다.
!python -c "import monai" || pip install -q -U "monai[nibabel, tqdm]"
!python -c "import wandb" || pip install -q -U wandb
import os
import numpy as np
from tqdm.auto import tqdm
import wandb
from monai.apps import DecathlonDataset
from monai.data import DataLoader, decollate_batch
from monai.losses import DiceLoss
from monai.inferers import sliding_window_inference
from monai.metrics import DiceMetric
from monai.networks.nets import SegResNet
from monai.transforms import (
Activations,
AsDiscrete,
Compose,
LoadImaged,
MapTransform,
NormalizeIntensityd,
Orientationd,
RandFlipd,
RandScaleIntensityd,
RandShiftIntensityd,
RandSpatialCropd,
Spacingd,
EnsureTyped,
EnsureChannelFirstd,
)
from monai.utils import set_determinism
import torch
다음으로 W&B를 사용하기 위해 Colab 인스턴스를 인증합니다.
wandb.login()
W&B Run 초기화
새로운 W&B run을 시작하여 실험 추적을 시작합니다.
wandb.init(project="monai-brain-tumor-segmentation")
적절한 구성 시스템을 사용하는 것이 재현 가능한 기계 학습을 위한 권장되는 모범 사례입니다. W&B를 사용하여 모든 실험에 대한 하이퍼파라미터를 추적할 수 있습니다.
config = wandb.config
config.seed = 0
config.roi_size = [224, 224, 144]
config.batch_size = 1
config.num_workers = 4
config.max_train_images_visualized = 20
config.max_val_images_visualized = 20
config.dice_loss_smoothen_numerator = 0
config.dice_loss_smoothen_denominator = 1e-5
config.dice_loss_squared_prediction = True
config.dice_loss_target_onehot = False
config.dice_loss_apply_sigmoid = True
config.initial_learning_rate = 1e-4
config.weight_decay = 1e-5
config.max_train_epochs = 50
config.validation_intervals = 1
config.dataset_dir = "./dataset/"
config.checkpoint_dir = "./checkpoints"
config.inference_roi_size = (128, 128, 64)
config.max_prediction_images_visualized = 20
결정론적 트레이닝을 활성화하거나 끄려면 모듈에 대한 임의 시드도 설정해야 합니다.
set_determinism(seed=config.seed)
# 디렉토리 생성
os.makedirs(config.dataset_dir, exist_ok=True)
os.makedirs(config.checkpoint_dir, exist_ok=True)
데이터 로딩 및 변환
여기서는 monai.transforms API를 사용하여 다중 클래스 레이블을 원-핫 형식의 다중 레이블 분할 작업으로 변환하는 사용자 정의 transform을 만듭니다.
class ConvertToMultiChannelBasedOnBratsClassesd(MapTransform):
"""
brats 클래스를 기반으로 레이블을 다중 채널로 변환합니다:
레이블 1은 종양 주위 부종입니다.
레이블 2는 GD-강화 종양입니다.
레이블 3은 괴사성 및 비강화 종양 코어입니다.
가능한 클래스는 TC(종양 코어), WT(전체 종양) 및 ET(강화 종양)입니다.
참조: https://github.com/Project-MONAI/tutorials/blob/main/3d_segmentation/brats_segmentation_3d.ipynb
"""
def __call__(self, data):
d = dict(data)
for key in self.keys:
result = []
# 레이블 2와 레이블 3을 병합하여 TC를 구성합니다.
result.append(torch.logical_or(d[key] == 2, d[key] == 3))
# 레이블 1, 2 및 3을 병합하여 WT를 구성합니다.
result.append(
torch.logical_or(
torch.logical_or(d[key] == 2, d[key] == 3), d[key] == 1
)
)
# 레이블 2는 ET입니다.
result.append(d[key] == 2)
d[key] = torch.stack(result, axis=0).float()
return d
다음으로 트레이닝 및 유효성 검사 데이터셋에 대한 transforms를 각각 설정합니다.
train_transform = Compose(
[
# 4개의 Nifti 이미지를 로드하고 함께 쌓습니다.
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys="image"),
EnsureTyped(keys=["image", "label"]),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(
keys=["image", "label"],
pixdim=(1.0, 1.0, 1.0),
mode=("bilinear", "nearest"),
),
RandSpatialCropd(
keys=["image", "label"], roi_size=config.roi_size, random_size=False
),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=0),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=1),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=2),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
RandScaleIntensityd(keys="image", factors=0.1, prob=1.0),
RandShiftIntensityd(keys="image", offsets=0.1, prob=1.0),
]
)
val_transform = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys="image"),
EnsureTyped(keys=["image", "label"]),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(
keys=["image", "label"],
pixdim=(1.0, 1.0, 1.0),
mode=("bilinear", "nearest"),
),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
]
)
데이터셋
이 실험에 사용된 데이터셋은 http://medicaldecathlon.com/에서 가져온 것입니다. 다중 모드 다중 사이트 MRI 데이터(FLAIR, T1w, T1gd, T2w)를 사용하여 신경교종, 괴사성/활성 종양 및 부종을 분할합니다. 데이터셋은 750개의 4D 볼륨(484 트레이닝 + 266 테스트)으로 구성됩니다.
DecathlonDataset을 사용하여 데이터셋을 자동으로 다운로드하고 추출합니다. MONAI CacheDataset을 상속하여 cache_num=N을 설정하여 트레이닝을 위해 N개의 항목을 캐시하고 메모리 크기에 따라 유효성 검사를 위해 모든 항목을 캐시하는 기본 인수를 사용할 수 있습니다.
train_dataset = DecathlonDataset(
root_dir=config.dataset_dir,
task="Task01_BrainTumour",
transform=val_transform,
section="training",
download=True,
cache_rate=0.0,
num_workers=4,
)
val_dataset = DecathlonDataset(
root_dir=config.dataset_dir,
task="Task01_BrainTumour",
transform=val_transform,
section="validation",
download=False,
cache_rate=0.0,
num_workers=4,
)
train_dataset에 train_transform을 적용하는 대신 트레이닝 및 유효성 검사 데이터셋 모두에 val_transform을 적용합니다. 이는 트레이닝 전에 데이터셋 분할의 샘플을 시각화하기 때문입니다.데이터셋 시각화
Weights & Biases는 이미지, 비디오, 오디오 등을 지원합니다. 다양한 미디어를 기록하여 결과를 탐색하고 run, 모델 및 데이터셋을 시각적으로 비교할 수 있습니다. 분할 마스크 오버레이 시스템을 사용하여 데이터 볼륨을 시각화합니다. 테이블에서 분할 마스크를 기록하려면 테이블의 각 행에 대해 wandb.Image 오브젝트를 제공해야 합니다.
아래 의사 코드에 예제가 제공되어 있습니다.
table = wandb.Table(columns=["ID", "Image"])
for id, img, label in zip(ids, images, labels):
mask_img = wandb.Image(
img,
masks={
"prediction": {"mask_data": label, "class_labels": class_labels}
# ...
},
)
table.add_data(id, img)
wandb.log({"Table": table})
이제 샘플 이미지, 레이블, wandb.Table 오브젝트 및 일부 관련 메타데이터를 가져와서 Weights & Biases 대시보드에 기록될 테이블의 행을 채우는 간단한 유틸리티 함수를 작성합니다.
def log_data_samples_into_tables(
sample_image: np.array,
sample_label: np.array,
split: str = None,
data_idx: int = None,
table: wandb.Table = None,
):
num_channels, _, _, num_slices = sample_image.shape
with tqdm(total=num_slices, leave=False) as progress_bar:
for slice_idx in range(num_slices):
ground_truth_wandb_images = []
for channel_idx in range(num_channels):
ground_truth_wandb_images.append(
masks = {
"ground-truth/Tumor-Core": {
"mask_data": sample_label[0, :, :, slice_idx],
"class_labels": {0: "background", 1: "Tumor Core"},
},
"ground-truth/Whole-Tumor": {
"mask_data": sample_label[1, :, :, slice_idx] * 2,
"class_labels": {0: "background", 2: "Whole Tumor"},
},
"ground-truth/Enhancing-Tumor": {
"mask_data": sample_label[2, :, :, slice_idx] * 3,
"class_labels": {0: "background", 3: "Enhancing Tumor"},
},
}
wandb.Image(
sample_image[channel_idx, :, :, slice_idx],
masks=masks,
)
)
table.add_data(split, data_idx, slice_idx, *ground_truth_wandb_images)
progress_bar.update(1)
return table
다음으로 wandb.Table 오브젝트와 데이터 시각화로 채울 수 있도록 구성되는 열을 정의합니다.
table = wandb.Table(
columns=[
"Split",
"Data Index",
"Slice Index",
"Image-Channel-0",
"Image-Channel-1",
"Image-Channel-2",
"Image-Channel-3",
]
)
그런 다음 각각 train_dataset 및 val_dataset을 반복하여 데이터 샘플에 대한 시각화를 생성하고 대시보드에 기록할 테이블의 행을 채웁니다.
# train_dataset에 대한 시각화 생성
max_samples = (
min(config.max_train_images_visualized, len(train_dataset))
if config.max_train_images_visualized > 0
else len(train_dataset)
)
progress_bar = tqdm(
enumerate(train_dataset[:max_samples]),
total=max_samples,
desc="Generating Train Dataset Visualizations:",
)
for data_idx, sample in progress_bar:
sample_image = sample["image"].detach().cpu().numpy()
sample_label = sample["label"].detach().cpu().numpy()
table = log_data_samples_into_tables(
sample_image,
sample_label,
split="train",
data_idx=data_idx,
table=table,
)
# val_dataset에 대한 시각화 생성
max_samples = (
min(config.max_val_images_visualized, len(val_dataset))
if config.max_val_images_visualized > 0
else len(val_dataset)
)
progress_bar = tqdm(
enumerate(val_dataset[:max_samples]),
total=max_samples,
desc="Generating Validation Dataset Visualizations:",
)
for data_idx, sample in progress_bar:
sample_image = sample["image"].detach().cpu().numpy()
sample_label = sample["label"].detach().cpu().numpy()
table = log_data_samples_into_tables(
sample_image,
sample_label,
split="val",
data_idx=data_idx,
table=table,
)
# 테이블을 대시보드에 기록
wandb.log({"Tumor-Segmentation-Data": table})
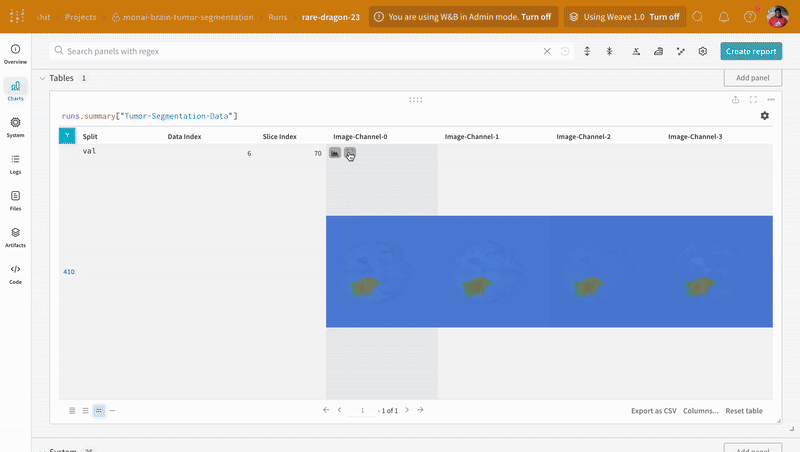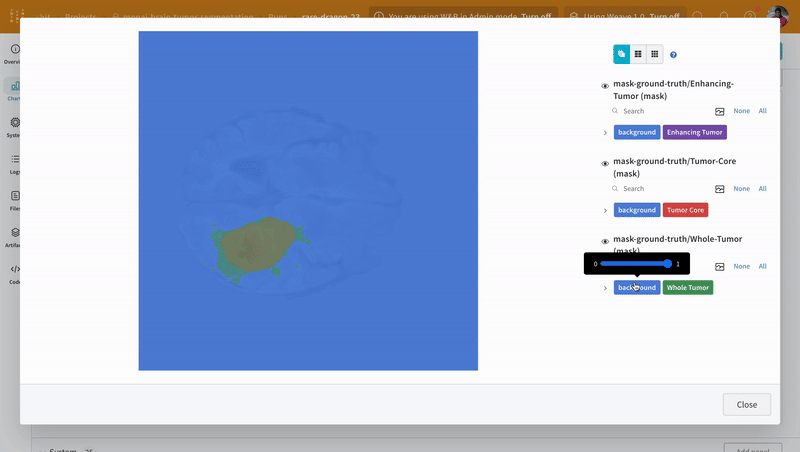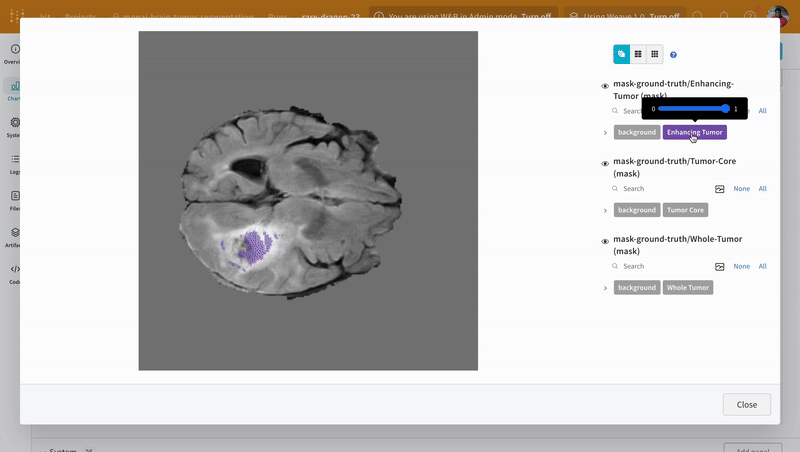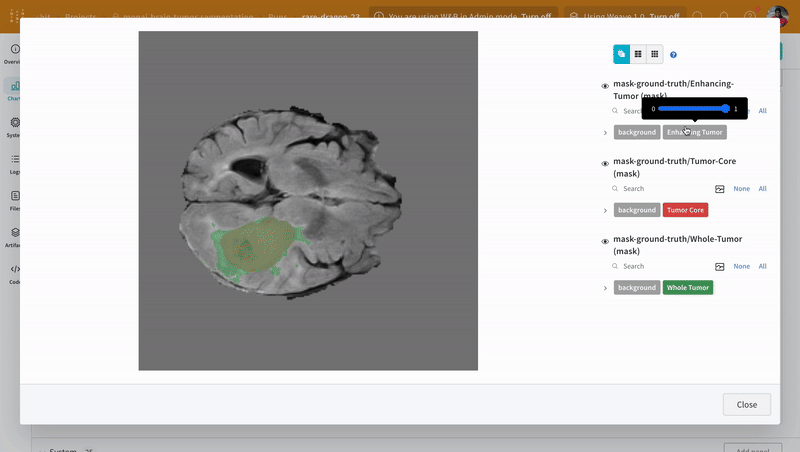
데이터는 대화형 테이블 형식으로 W&B 대시보드에 나타납니다. 각 행에서 데이터 볼륨의 특정 슬라이스의 각 채널이 해당 분할 마스크로 오버레이된 것을 볼 수 있습니다. Weave 쿼리를 작성하여 테이블의 데이터를 필터링하고 특정 행에 집중할 수 있습니다.
 |
|---|
| 기록된 테이블 데이터의 예. |
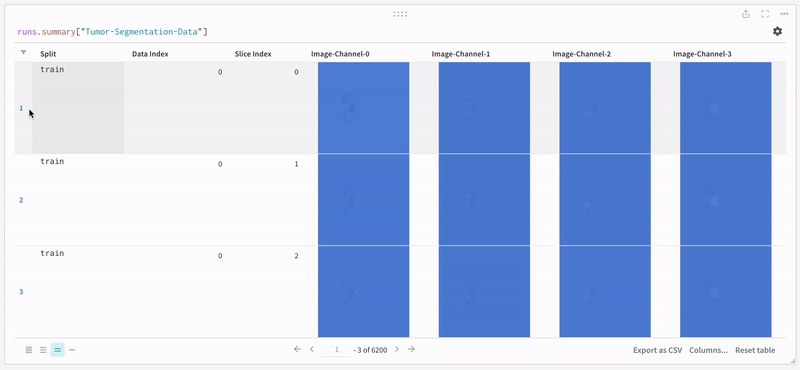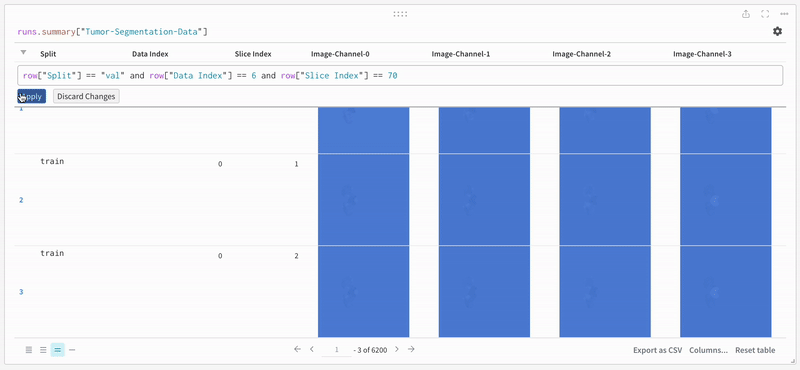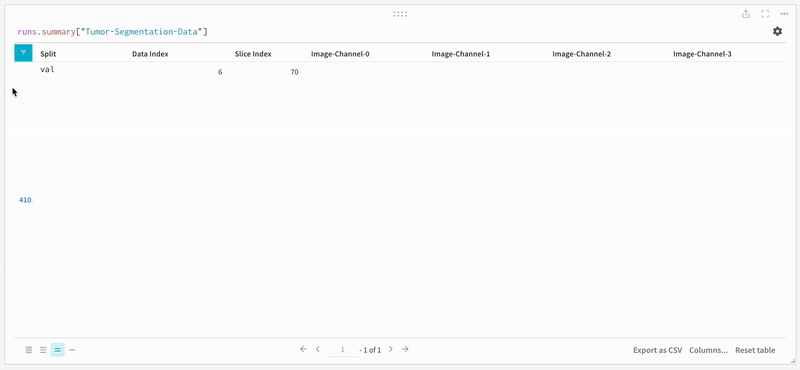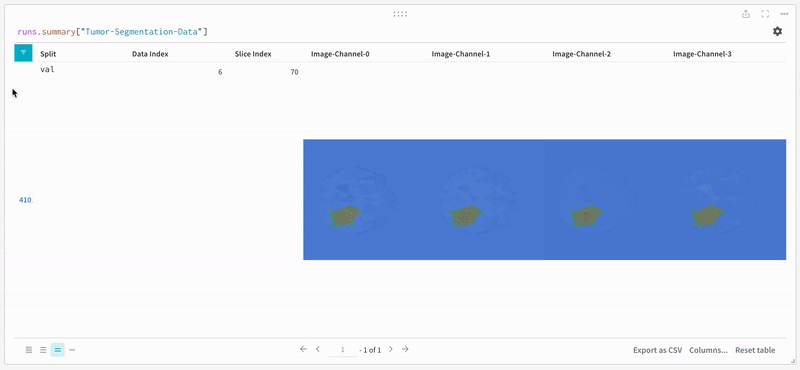
이미지를 열고 대화형 오버레이를 사용하여 각 분할 마스크와 상호 작용하는 방법을 확인합니다.
 |
|---|
| 시각화된 분할 맵의 예. |
데이터 로딩
데이터셋에서 데이터를 로드하기 위한 PyTorch DataLoaders를 만듭니다. DataLoaders를 만들기 전에 트레이닝을 위해 데이터를 사전 처리하고 변환하기 위해 train_dataset에 대한 transform을 train_transform으로 설정합니다.
# 트레이닝 데이터셋에 train_transforms 적용
train_dataset.transform = train_transform
# train_loader 생성
train_loader = DataLoader(
train_dataset,
batch_size=config.batch_size,
shuffle=True,
num_workers=config.num_workers,
)
# val_loader 생성
val_loader = DataLoader(
val_dataset,
batch_size=config.batch_size,
shuffle=False,
num_workers=config.num_workers,
)
모델, 손실 및 옵티마이저 생성
이 튜토리얼에서는 자동 인코더 정규화를 사용한 3D MRI 뇌종양 분할 논문을 기반으로 SegResNet 모델을 만듭니다. SegResNet 모델은 monai.networks API의 일부로 PyTorch 모듈로 구현되었으며 옵티마이저 및 학습률 스케줄러도 함께 제공됩니다.
device = torch.device("cuda:0")
# 모델 생성
model = SegResNet(
blocks_down=[1, 2, 2, 4],
blocks_up=[1, 1, 1],
init_filters=16,
in_channels=4,
out_channels=3,
dropout_prob=0.2,
).to(device)
# 옵티마이저 생성
optimizer = torch.optim.Adam(
model.parameters(),
config.initial_learning_rate,
weight_decay=config.weight_decay,
)
# 학습률 스케줄러 생성
lr_scheduler = torch.optim.lr_scheduler.CosineAnnealingLR(
optimizer, T_max=config.max_train_epochs
)
monai.losses API를 사용하여 손실을 다중 레이블 DiceLoss로 정의하고 monai.metrics API를 사용하여 해당 주사위 메트릭을 정의합니다.
loss_function = DiceLoss(
smooth_nr=config.dice_loss_smoothen_numerator,
smooth_dr=config.dice_loss_smoothen_denominator,
squared_pred=config.dice_loss_squared_prediction,
to_onehot_y=config.dice_loss_target_onehot,
sigmoid=config.dice_loss_apply_sigmoid,
)
dice_metric = DiceMetric(include_background=True, reduction="mean")
dice_metric_batch = DiceMetric(include_background=True, reduction="mean_batch")
post_trans = Compose([Activations(sigmoid=True), AsDiscrete(threshold=0.5)])
# 자동 혼합 정밀도를 사용하여 트레이닝 가속화
scaler = torch.cuda.amp.GradScaler()
torch.backends.cudnn.benchmark = True
혼합 정밀도 추론을 위한 작은 유틸리티를 정의합니다. 이는 트레이닝 프로세스의 유효성 검사 단계와 트레이닝 후 모델을 실행하려는 경우에 유용합니다.
def inference(model, input):
def _compute(input):
return sliding_window_inference(
inputs=input,
roi_size=(240, 240, 160),
sw_batch_size=1,
predictor=model,
overlap=0.5,
)
with torch.cuda.amp.autocast():
return _compute(input)
트레이닝 및 유효성 검사
트레이닝 전에 트레이닝 및 유효성 검사 실험을 추적하기 위해 나중에 wandb.log()로 기록될 메트릭 속성을 정의합니다.
wandb.define_metric("epoch/epoch_step")
wandb.define_metric("epoch/*", step_metric="epoch/epoch_step")
wandb.define_metric("batch/batch_step")
wandb.define_metric("batch/*", step_metric="batch/batch_step")
wandb.define_metric("validation/validation_step")
wandb.define_metric("validation/*", step_metric="validation/validation_step")
batch_step = 0
validation_step = 0
metric_values = []
metric_values_tumor_core = []
metric_values_whole_tumor = []
metric_values_enhanced_tumor = []
표준 PyTorch 트레이닝 루프 실행
# W&B Artifact 오브젝트 정의
artifact = wandb.Artifact(
name=f"{wandb.run.id}-checkpoint", type="model"
)
epoch_progress_bar = tqdm(range(config.max_train_epochs), desc="Training:")
for epoch in epoch_progress_bar:
model.train()
epoch_loss = 0
total_batch_steps = len(train_dataset) // train_loader.batch_size
batch_progress_bar = tqdm(train_loader, total=total_batch_steps, leave=False)
# 트레이닝 단계
for batch_data in batch_progress_bar:
inputs, labels = (
batch_data["image"].to(device),
batch_data["label"].to(device),
)
optimizer.zero_grad()
with torch.cuda.amp.autocast():
outputs = model(inputs)
loss = loss_function(outputs, labels)
scaler.scale(loss).backward()
scaler.step(optimizer)
scaler.update()
epoch_loss += loss.item()
batch_progress_bar.set_description(f"train_loss: {loss.item():.4f}:")
## 배치별 트레이닝 손실을 W&B에 기록
wandb.log({"batch/batch_step": batch_step, "batch/train_loss": loss.item()})
batch_step += 1
lr_scheduler.step()
epoch_loss /= total_batch_steps
## 배치별 트레이닝 손실과 학습률을 W&B에 기록
wandb.log(
{
"epoch/epoch_step": epoch,
"epoch/mean_train_loss": epoch_loss,
"epoch/learning_rate": lr_scheduler.get_last_lr()[0],
}
)
epoch_progress_bar.set_description(f"Training: train_loss: {epoch_loss:.4f}:")
# 유효성 검사 및 모델 체크포인트 단계
if (epoch + 1) % config.validation_intervals == 0:
model.eval()
with torch.no_grad():
for val_data in val_loader:
val_inputs, val_labels = (
val_data["image"].to(device),
val_data["label"].to(device),
)
val_outputs = inference(model, val_inputs)
val_outputs = [post_trans(i) for i in decollate_batch(val_outputs)]
dice_metric(y_pred=val_outputs, y=val_labels)
dice_metric_batch(y_pred=val_outputs, y=val_labels)
metric_values.append(dice_metric.aggregate().item())
metric_batch = dice_metric_batch.aggregate()
metric_values_tumor_core.append(metric_batch[0].item())
metric_values_whole_tumor.append(metric_batch[1].item())
metric_values_enhanced_tumor.append(metric_batch[2].item())
dice_metric.reset()
dice_metric_batch.reset()
checkpoint_path = os.path.join(config.checkpoint_dir, "model.pth")
torch.save(model.state_dict(), checkpoint_path)
# W&B Artifacts를 사용하여 모델 체크포인트를 기록하고 버전 관리합니다.
artifact.add_file(local_path=checkpoint_path)
wandb.log_artifact(artifact, aliases=[f"epoch_{epoch}"])
# 유효성 검사 메트릭을 W&B 대시보드에 기록합니다.
wandb.log(
{
"validation/validation_step": validation_step,
"validation/mean_dice": metric_values[-1],
"validation/mean_dice_tumor_core": metric_values_tumor_core[-1],
"validation/mean_dice_whole_tumor": metric_values_whole_tumor[-1],
"validation/mean_dice_enhanced_tumor": metric_values_enhanced_tumor[-1],
}
)
validation_step += 1
# 이 Artifact가 기록을 마칠 때까지 기다립니다.
artifact.wait()
wandb.log로 코드를 계측하면 트레이닝 및 유효성 검사 프로세스와 관련된 모든 메트릭뿐만 아니라 W&B 대시보드의 모든 시스템 메트릭(이 경우 CPU 및 GPU)을 추적할 수 있습니다.
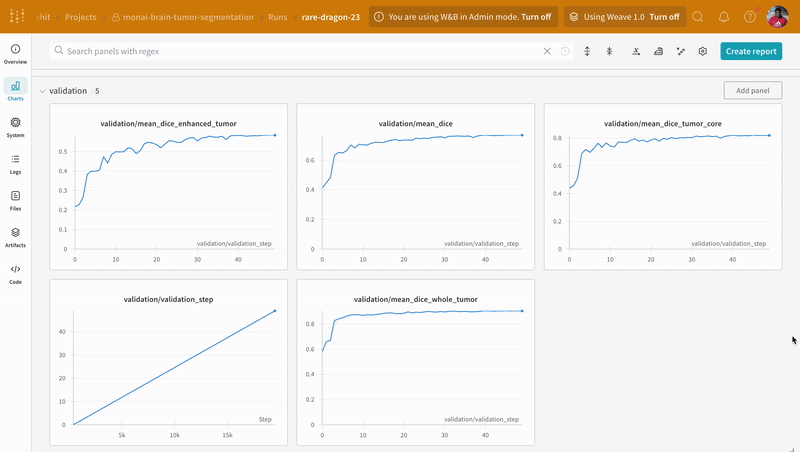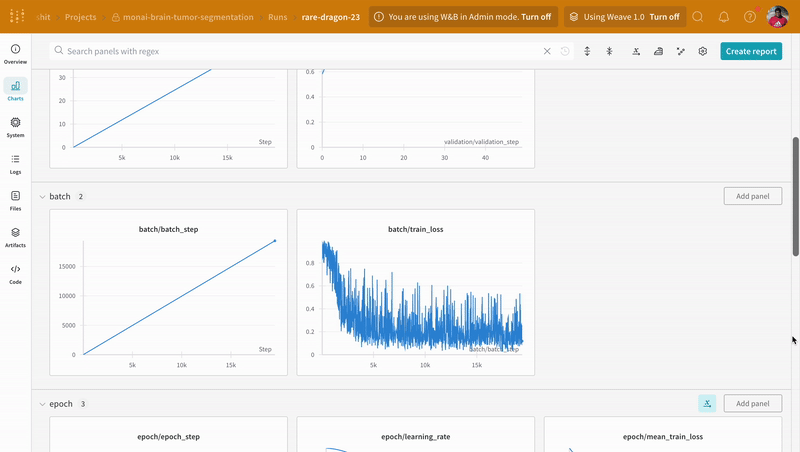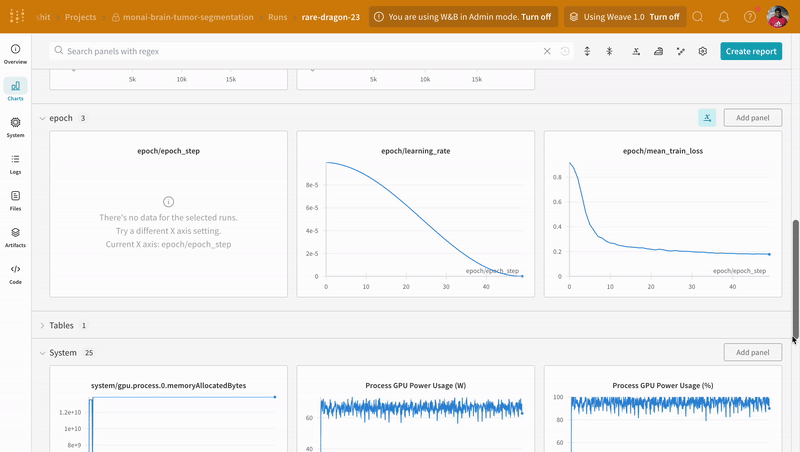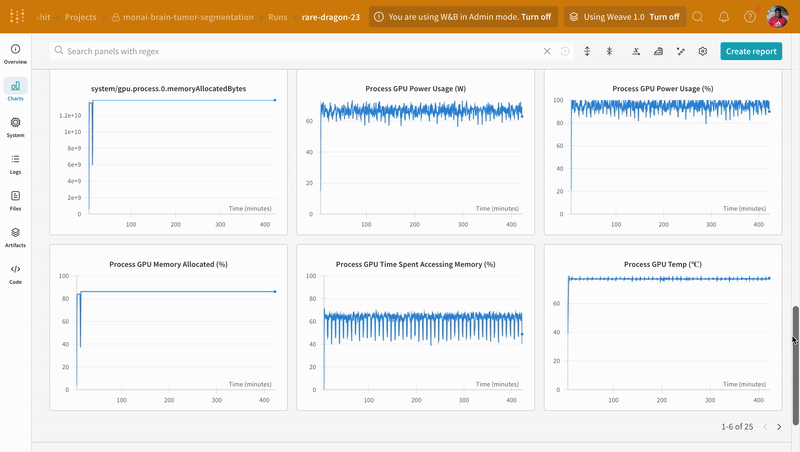 |
|---|
| W&B에서 트레이닝 및 유효성 검사 프로세스 추적의 예. |
트레이닝 중에 기록된 모델 체크포인트 Artifacts의 다른 버전에 액세스하려면 W&B run 대시보드의 Artifacts 탭으로 이동합니다.
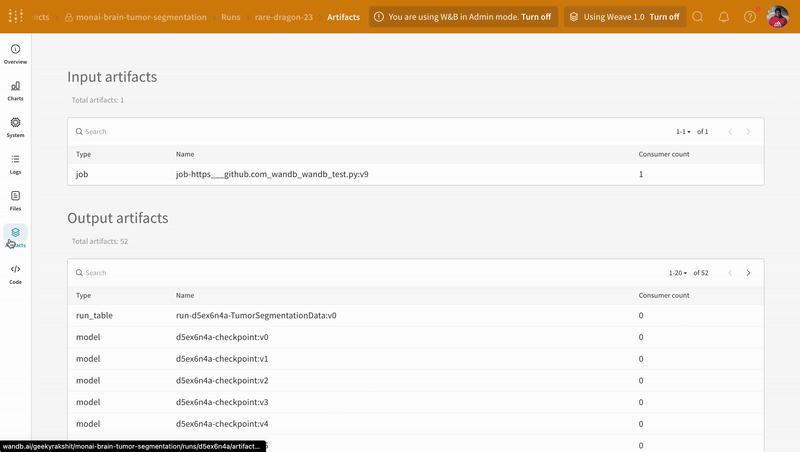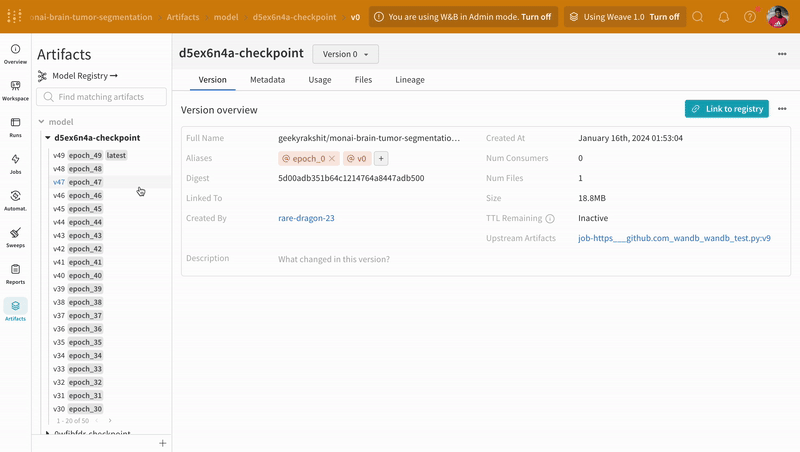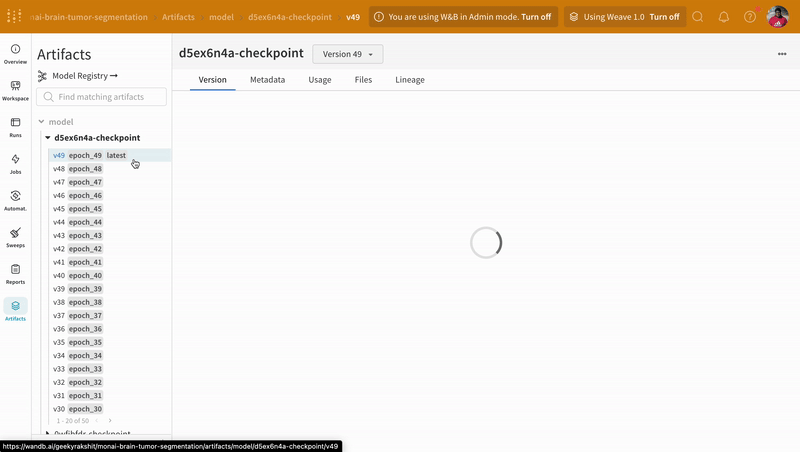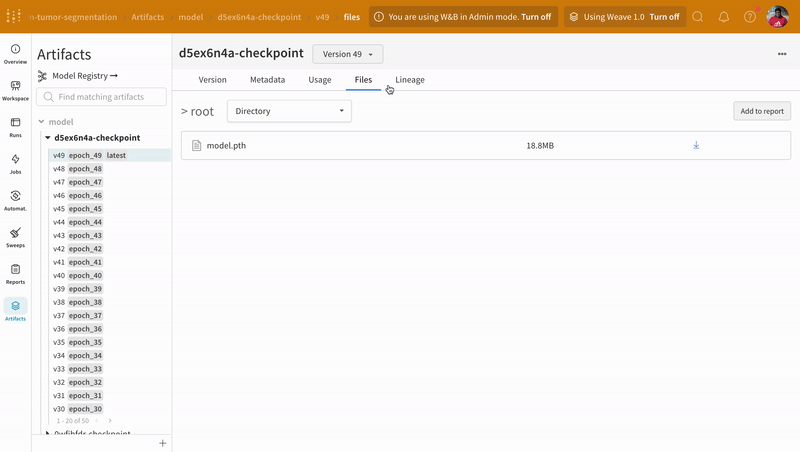 |
|---|
| W&B에서 모델 체크포인트 로깅 및 버전 관리의 예. |
추론
Artifacts 인터페이스를 사용하여 평균 에포크별 트레이닝 손실인 Artifact의 어떤 버전이 가장 적합한 모델 체크포인트인지 선택할 수 있습니다. Artifact의 전체 계보를 탐색하고 필요한 버전을 사용할 수도 있습니다.
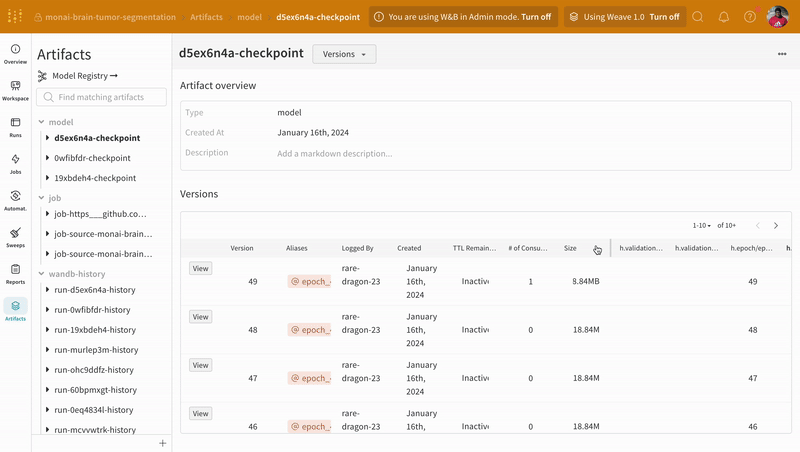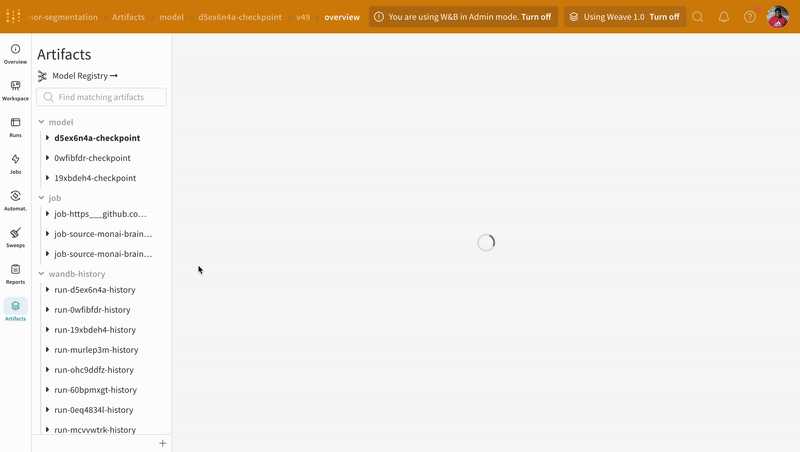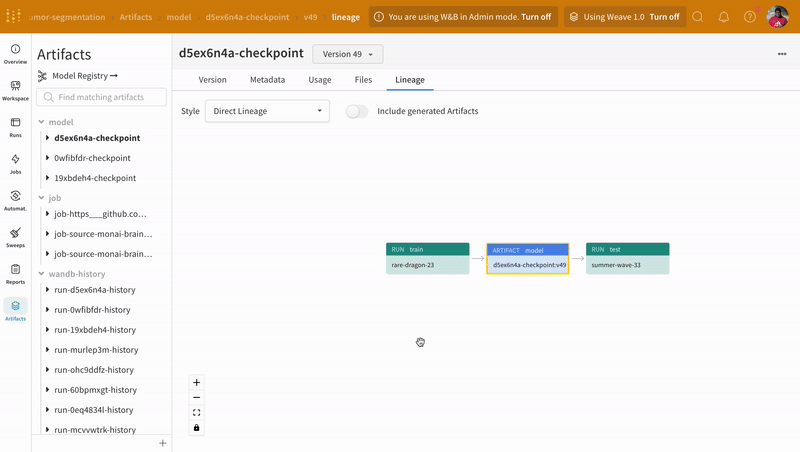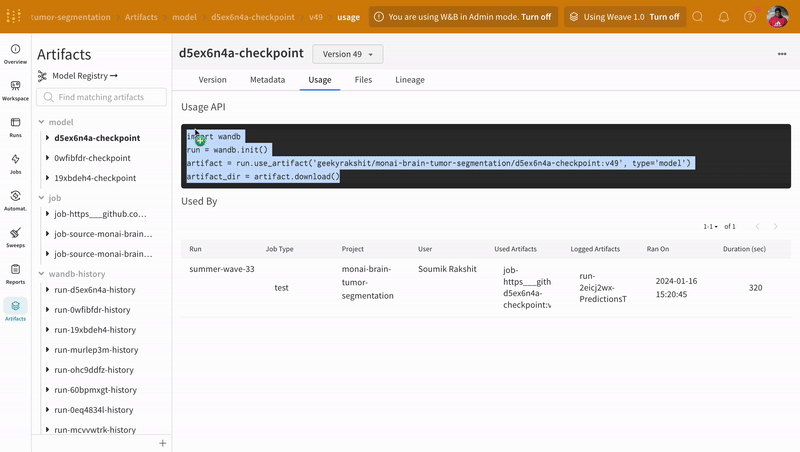 |
|---|
| W&B에서 모델 Artifact 추적의 예. |
최고의 에포크별 평균 트레이닝 손실을 가진 모델 Artifact의 버전을 가져오고 체크포인트 상태 사전을 모델에 로드합니다.
model_artifact = wandb.use_artifact(
"geekyrakshit/monai-brain-tumor-segmentation/d5ex6n4a-checkpoint:v49",
type="model",
)
model_artifact_dir = model_artifact.download()
model.load_state_dict(torch.load(os.path.join(model_artifact_dir, "model.pth")))
model.eval()
예측 시각화 및 그라운드 트루스 레이블과 비교
대화형 분할 마스크 오버레이를 사용하여 사전 트레이닝된 모델의 예측을 시각화하고 해당 그라운드 트루스 분할 마스크와 비교하는 또 다른 유틸리티 함수를 만듭니다.
def log_predictions_into_tables(
sample_image: np.array,
sample_label: np.array,
predicted_label: np.array,
split: str = None,
data_idx: int = None,
table: wandb.Table = None,
):
num_channels, _, _, num_slices = sample_image.shape
with tqdm(total=num_slices, leave=False) as progress_bar:
for slice_idx in range(num_slices):
wandb_images = []
for channel_idx in range(num_channels):
wandb_images += [
wandb.Image(
sample_image[channel_idx, :, :, slice_idx],
masks={
"ground-truth/Tumor-Core": {
"mask_data": sample_label[0, :, :, slice_idx],
"class_labels": {0: "background", 1: "Tumor Core"},
},
"prediction/Tumor-Core": {
"mask_data": predicted_label[0, :, :, slice_idx] * 2,
"class_labels": {0: "background", 2: "Tumor Core"},
},
},
),
wandb.Image(
sample_image[channel_idx, :, :, slice_idx],
masks={
"ground-truth/Whole-Tumor": {
"mask_data": sample_label[1, :, :, slice_idx],
"class_labels": {0: "background", 1: "Whole Tumor"},
},
"prediction/Whole-Tumor": {
"mask_data": predicted_label[1, :, :, slice_idx] * 2,
"class_labels": {0: "background", 2: "Whole Tumor"},
},
},
),
wandb.Image(
sample_image[channel_idx, :, :, slice_idx],
masks={
"ground-truth/Enhancing-Tumor": {
"mask_data": sample_label[2, :, :, slice_idx],
"class_labels": {0: "background", 1: "Enhancing Tumor"},
},
"prediction/Enhancing-Tumor": {
"mask_data": predicted_label[2, :, :, slice_idx] * 2,
"class_labels": {0: "background", 2: "Enhancing Tumor"},
},
},
),
]
table.add_data(split, data_idx, slice_idx, *wandb_images)
progress_bar.update(1)
return table
예측 결과를 예측 테이블에 기록합니다.
# 예측 테이블 생성
prediction_table = wandb.Table(
columns=[
"Split",
"Data Index",
"Slice Index",
"Image-Channel-0/Tumor-Core",
"Image-Channel-1/Tumor-Core",
"Image-Channel-2/Tumor-Core",
"Image-Channel-3/Tumor-Core",
"Image-Channel-0/Whole-Tumor",
"Image-Channel-1/Whole-Tumor",
"Image-Channel-2/Whole-Tumor",
"Image-Channel-3/Whole-Tumor",
"Image-Channel-0/Enhancing-Tumor",
"Image-Channel-1/Enhancing-Tumor",
"Image-Channel-2/Enhancing-Tumor",
"Image-Channel-3/Enhancing-Tumor",
]
)
# 추론 및 시각화 수행
with torch.no_grad():
config.max_prediction_images_visualized
max_samples = (
min(config.max_prediction_images_visualized, len(val_dataset))
if config.max_prediction_images_visualized > 0
else len(val_dataset)
)
progress_bar = tqdm(
enumerate(val_dataset[:max_samples]),
total=max_samples,
desc="Generating Predictions:",
)
for data_idx, sample in progress_bar:
val_input = sample["image"].unsqueeze(0).to(device)
val_output = inference(model, val_input)
val_output = post_trans(val_output[0])
prediction_table = log_predictions_into_tables(
sample_image=sample["image"].cpu().numpy(),
sample_label=sample["label"].cpu().numpy(),
predicted_label=val_output.cpu().numpy(),
data_idx=data_idx,
split="validation",
table=prediction_table,
)
wandb.log({"Predictions/Tumor-Segmentation-Data": prediction_table})
# 실험 종료
wandb.finish()
대화형 분할 마스크 오버레이를 사용하여 각 클래스에 대한 예측된 분할 마스크와 그라운드 트루스 레이블을 분석하고 비교합니다.
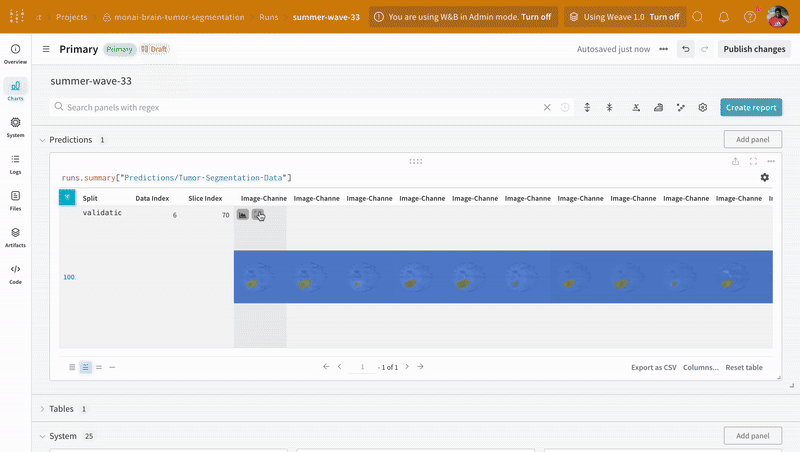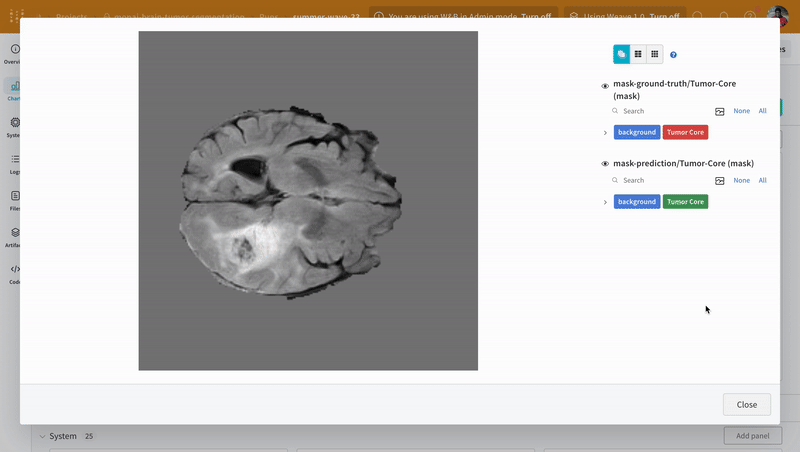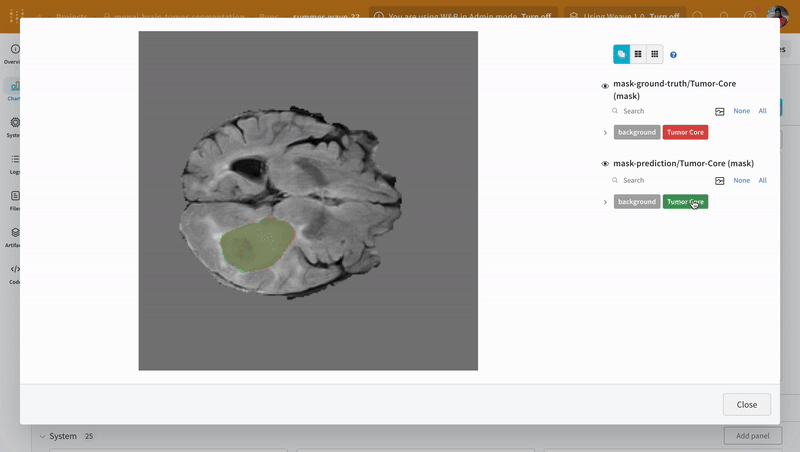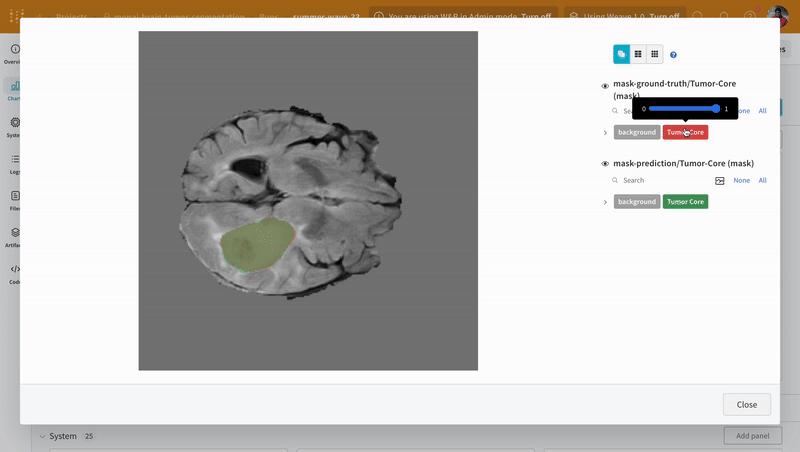 |
|---|
| W&B에서 예측 및 그라운드 트루스 시각화의 예. |
감사의 말씀 및 추가 자료
[i18n] feedback_title
[i18n] feedback_question
Glad to hear it! Please tell us how we can improve.
Sorry to hear that. Please tell us how we can improve.